Kit FastKing RT (avec gDNase)
Caractéristiques
■ Haute efficacité : L'enzyme FastKing RT est modifiée avec un motif hydrophobe, avec une efficacité RT supérieure à 95 %.
■ Sensible : aussi peu que 1 ng de modèles peuvent être identifiés avec précision.
■ Résistance : Capable de transcription inverse de modèles complexes, avec une parfaite résistance aux impuretés.
■ Flexible : l'élimination de l'ADN génomique et la transcription inverse ont été effectuées séparément. Les amorces ont été mélangées séparément dans un tube, de manière flexible pour changer d'autres amorces.
spécification
Type : transcriptase inverse modifiée par un gène, gDNase
Procédures : en deux étapes (élimination de l'ADN génomique et RT)
Efficacité RT : > 95 %
Gabarit : 1 ng - 2 g
Temps de fonctionnement : ~21 min
Applications : L'ADNc transcrit en sens inverse peut être utilisé dans la PCR conventionnelle, la PCR en temps réel, la construction de bibliothèques d'ADNc.
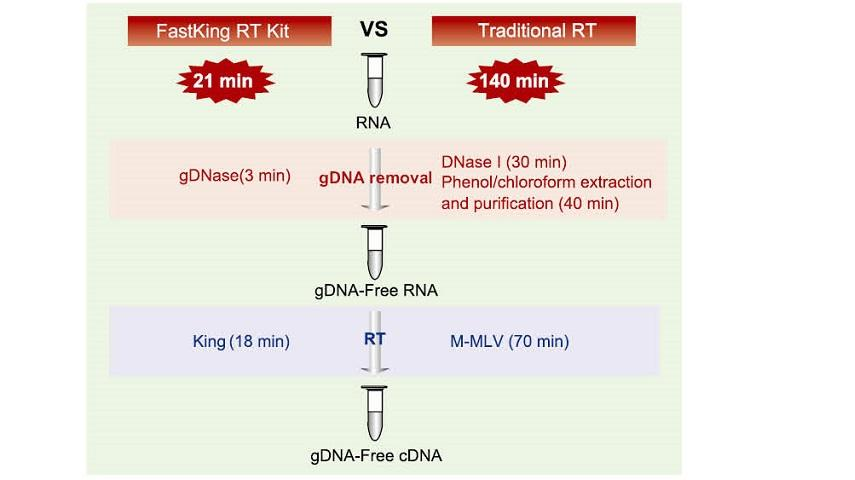
21 min de réaction dans un tube
Il ne faut que 21 minutes pour terminer l'élimination de l'ADNg et le processus efficace de transcription inverse dans le même tube sans remplacer le tube de réaction et le processus de traitement indépendant à la DNase I. Par rapport à la méthode traditionnelle qui nécessite un fonctionnement en 12 étapes et une réaction de 140 minutes, elle simplifie considérablement les étapes de fonctionnement et permet de gagner beaucoup de temps.
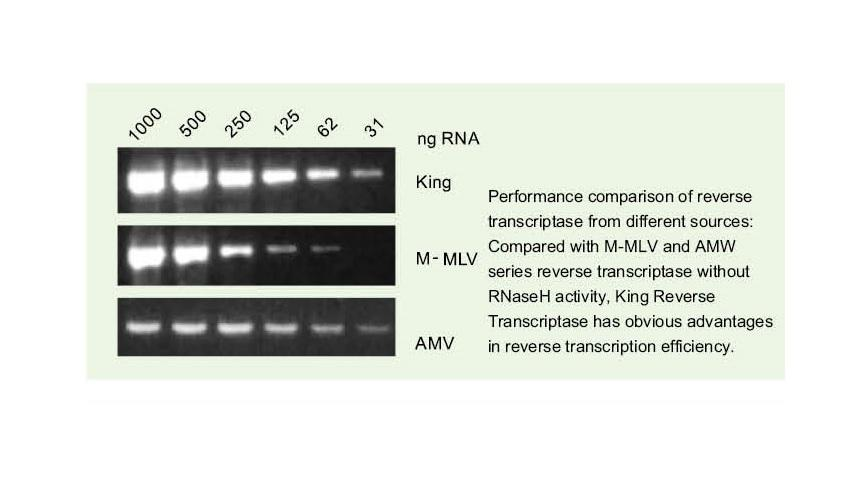
Qualité exceptionnelle de King RTase
—— Efficacité de transcription inverse ultra-élevée
—— L'efficacité de la transcription inverse est supérieure à 95%
La transcriptase inverse générale a une efficacité de transcription inverse de 40 à 60 %, et le rendement en ADNc peut être augmenté par une quantité de charge d'ARN plus élevée. La transcriptase inverse King peut atteindre une efficacité de transcription inverse de plus de 95 % en raison de sa haute affinité unique pour les matrices d'ARN. Par conséquent, les expériences ultérieures peuvent être satisfaites sans avoir besoin d'une grande quantité d'entrée d'ARN, ce qui économise l'ARN et permet une pureté élevée et un rendement élevé d'ADNc.
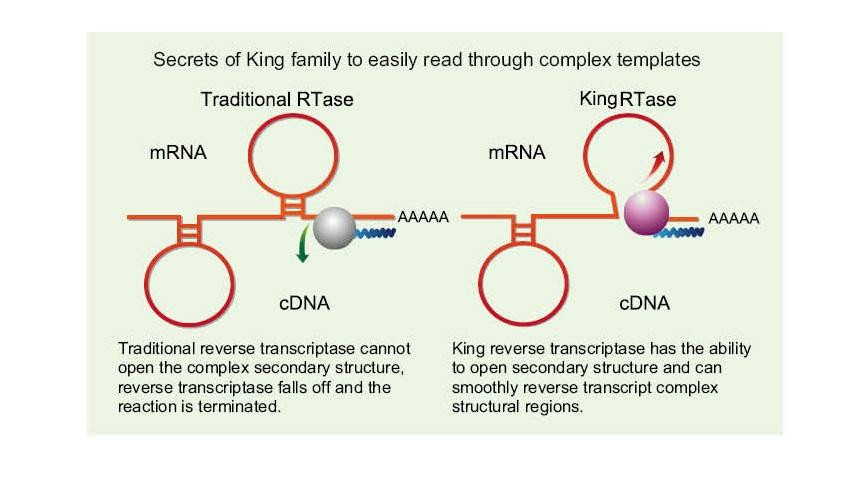
Lisez facilement des modèles complexes
——Lire facilement à travers des modèles à GC élevé et complexes
L'ARN simple brin a une large gamme de régions de structure secondaire complexes en raison de la liaison hydrogène entre les brins. La transcriptase inverse ordinaire peut conduire à l'arrêt de la transcription inverse lors de la rencontre d'une structure secondaire complexe, donc incapable de terminer avec succès la synthèse d'ADNc. Cependant, la nouvelle génération de transcriptase inverse King possède un domaine structurel unique, qui peut détruire la liaison hydrogène entre les brins d'ARN, ouvrant ainsi la structure secondaire complexe de l'ARN et assurant la transcription inverse en douceur.
Tous les produits peuvent être personnalisés pour ODM/OEM. Pour plus de détails,s'il vous plaît cliquez sur Service personnalisé (ODM/OEM)
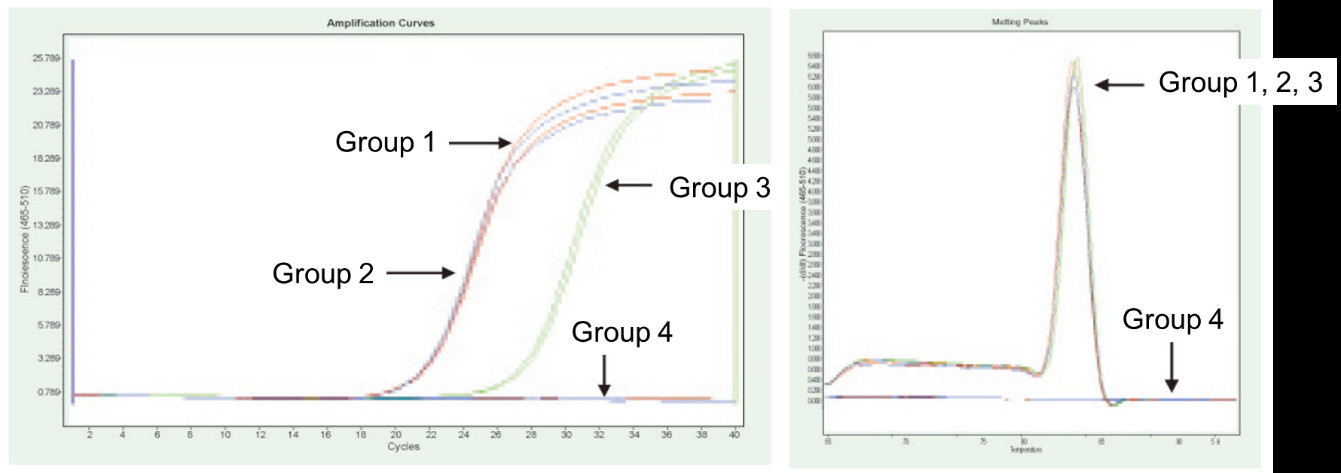 |
Groupe 1 : transcription inverse sans traitement à la gDNase ; Groupe 2 : Pas de traitement à la gDNase et pas de transcription inverse ; Groupe 3 : transcription inverse après traitement à la gDNase ; Groupe 4 : traitement à la gDNase sans transcription inverse. Méthodes : Détection par PCR quantitative par fluorescence du gène TNF-alpha (amorce conçue sur un exon avec un ADNc ou un génome comme matrice) en utilisant 1 g d'ARN de cellules Hela (avec un résidu génomique) comme matrice. Résultats : comme le montre la figure, le groupe 2 peut refléter le résidu du génome dans l'ARN, le groupe 3 peut refléter avec précision le véritable niveau d'expression du TNF-alpha, le groupe 1 a des erreurs dans les résultats quantitatifs finaux en raison du résidu du génome, et le groupe 4 montre que le kit FastKing RT peut éliminer complètement l'ADN génomique résiduel dans ARN. |
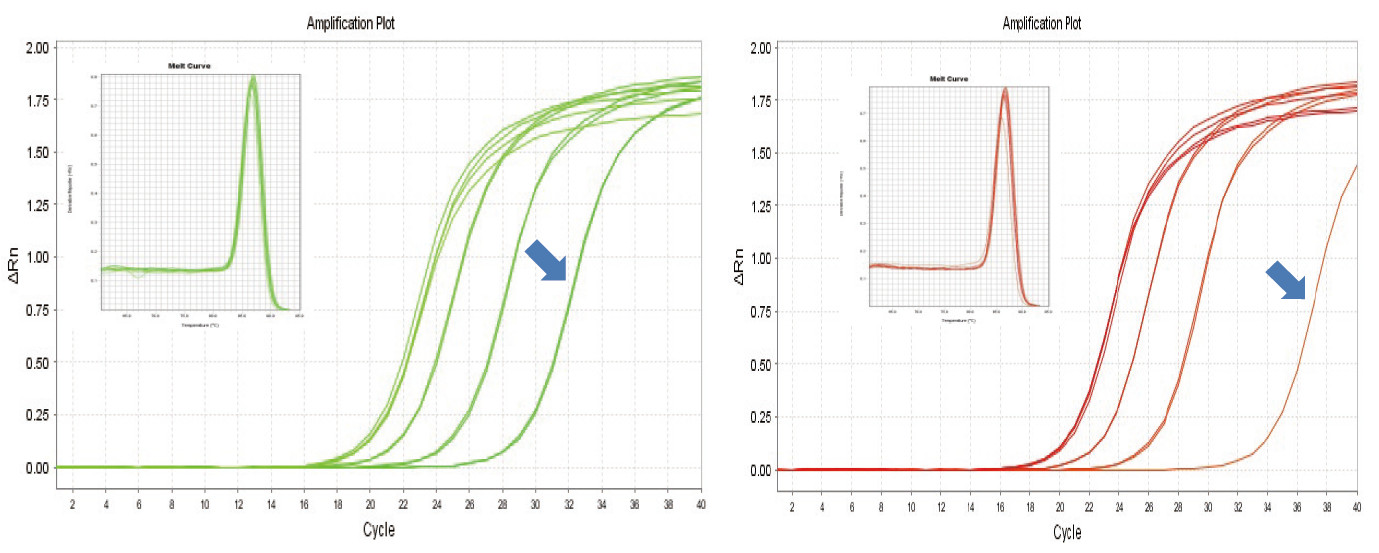 |
Figure 1. La transcription inverse de l'ARN de souris a été réalisée à l'aide du kit TIANGEN FastKing RT (à gauche) et du produit pertinent du fournisseur A (à droite), puis le gène MM5 a été amplifié de manière quantitative à l'aide de TIANGEN SuperReal PreMix Plus (SYBR Green). La courbe d'amplification et la courbe de fusion ont été analysées. L'entrée d'ARN était respectivement de 1000 ng, 100 ng, 10 ng et 1 ng. Les résultats montrent que le kit TIANGEN FastKing RT a un gradient de transcription inverse clair et une faible valeur Ct, et présente des avantages évidents pour la transcription inverse d'une matrice de faible abondance (1 ng, flèche bleue). |
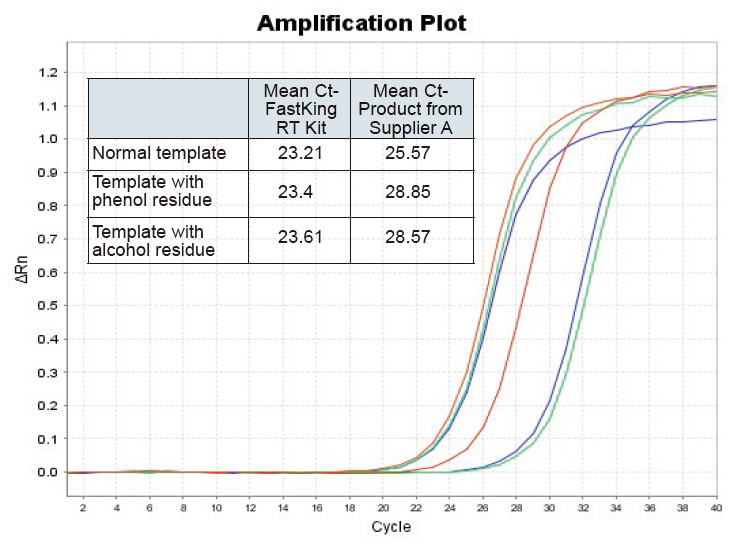 |
Figure 2. Transcription inverse du modèle d'ARN normal (rouge), modèle avec un grand résidu de phénol (vert) et modèle avec un résidu d'alcool (bleu) de rats à l'aide du kit TIANGEN FastKing RT et du produit pertinent du fournisseur A respectivement, quantifier les gènes RNC à l'aide de TIANGEN SuperReal PreMix Plus (SYBR Green), les courbes d'amplification et les valeurs Ct ont été analysées. Les résultats montrent que le kit TIANGEN FastKing RT a la valeur Ct quantitative la plus faible après transcription inverse et une excellente résistance au stress, et présente des avantages évidents pour les modèles avec des résidus d'impuretés élevés |
L'ARN A-1 est dégradé
—— Purifier l'ARN de haute qualité sans contamination. Le matériel à partir duquel l'ARN est extrait doit être aussi frais que possible pour empêcher la dégradation de l'ARN. Analyser l'intégrité de l'ARN sur gel dénaturé avant la réaction RT. Après extraction de l'ARN, il doit être conservé dans du formamide à 100 %. Si un inhibiteur de RNase est utilisé, la température de chauffage doit être < 45 °C et le pH doit être inférieur à 8,0, sinon l'inhibiteur libérera toute la RNase liée. De plus, l'inhibiteur de RNase doit être ajouté dans des solutions contenant ≥ 0,8 mM de DTT.
L'ARN A-2 contient des inhibiteurs des réactions de transcription inverse
——Les inhibiteurs de transcription inverse comprennent le SDS, l'EDTA, le glycérol, le pyrophosphate de sodium, la spermidine, le formamide, le sel de guanidine, etc. Mélangez l'ARN de contrôle avec l'échantillon et comparez le rendement avec la réaction de l'ARN de contrôle pour vérifier s'il existe un inhibiteur. Laver la précipitation de l'ARN avec de l'éthanol à 70 % (v/v) pour éliminer les inhibiteurs.
A-3 Annelage insuffisant des amorces utilisées pour synthétiser le premier brin d'ADNc
——Déterminer que la température de recuit convient aux amorces utilisées dans l'expérience. Pour les hexamères aléatoires, il est recommandé de maintenir la température à 25°C pendant 10 min avant d'atteindre la température de réaction. Pour les amorces spécifiques au gène (GSP), essayez d'autres GSP ou passez à l'oligo(dT) ou à l'hexamère aléatoire.
A-4 Petite quantité d'ARN de départ
——Augmenter la quantité d'ARN. Pour les échantillons d'ARN inférieurs à 50 ng, 0,1 g à 0,5 g d'acétyl BSA peuvent être utilisés dans la synthèse du premier brin d'ADNc
A-5 La séquence cible n'est pas exprimée dans les tissus analysés.
——Essayez d'autres tissus.
La réaction PCR A-6 échoue
——Pour la RT-PCR en deux étapes, la matrice d'ADNc dans l'étape de PCR ne peut pas dépasser 1/5 du volume de réaction.
A-1 annelage non spécifique des amorces et des matrices
—— L'extrémité 3' des amorces ne doit pas contenir 2-3 dG ou dC. Utilisez des amorces spécifiques au gène dans la synthèse du premier brin au lieu d'amorces aléatoires ou d'oligo(dT). Utilisez une température de recuit plus élevée dans les premiers cycles, puis une température de recuit plus basse. Utilisez l'ADN polymérase Taq à démarrage à chaud pour la PCR afin d'améliorer la spécificité de la réaction.
A-2 Mauvaise conception des amorces spécifiques d'un gène
——Suivre les mêmes principes pour la conception des amorces d'amplification.
ARN A-3 contaminé par de l'ADN génomique
——Traiter l'ARN avec de la DNase I de qualité PCR. Mettre en place une réaction de contrôle sans transcription inverse pour détecter la contamination de l'ADN.
A-4 Formage du dimère d'amorce
——Concevoir des amorces sans séquences complémentaires à l'extrémité 3'.
A-5 Mg trop élevé2+ concentration
——Optimiser le magnésium2+ concentration pour chaque combinaison de matrice et d'amorce
A-6 Contaminé par de l'ADN étranger
——Utilisez des pointes résistantes aux aérosols et des enzymes UDG.
A-1 Le contenu du produit du premier brin est trop élevé
——Réduire la quantité de produit du premier brin dans l'étape de réaction PCR classique.
A-2 Quantité d'amorce trop élevée dans la réaction PCR
——Réduire l'entrée d'amorce.
A-3 Trop de cycles
——Optimiser les conditions de réaction PCR et réduire le nombre de cycles PCR.
A-4 Température de recuit trop basse
——Augmenter la température de recuit pour éviter une initiation et une extension non spécifiques.
A-5 Amplification non spécifique de fragments d'oligonucléotides générés par la dégradation de l'ADN par la DNase ——Extraire de l'ARN de haute qualité pour éviter la contamination de l'ADN.
La RT-PCR consiste à transcrire à l'envers l'ARN en ADNc, puis à utiliser l'ADNc à transcrit à l'envers comme matrice pour la réaction PCR afin d'amplifier le fragment cible. Choisissez soit des amorces aléatoires, soit des amorces Oligo dT et des amorces spécifiques au gène en fonction des conditions spécifiques de l'expérience. Toutes les amorces ci-dessus peuvent être utilisées pour l'ARNm de cellules eucaryotes courtes sans structure en épingle à cheveux.
Amorce aléatoire : Convient aux ARN longs à structure en épingle à cheveux, ainsi qu'à toutes sortes d'ARN tels que l'ARNr, l'ARNm, l'ARNt, etc. Ils sont principalement utilisés pour la réaction RT-PCR d'une matrice unique.
Oligo dT : Convient pour l'ARN avec une queue PolyA (l'ARN procaryote, l'ARNr et l'ARNt Oligo dT eucaryotes n'ont pas de queue PolyA). Parce que Oligo dT est lié à la queue PolyA, la qualité des échantillons d'ARN doit être élevée, et même une petite quantité de dégradation réduira considérablement la quantité de synthèse d'ADNc pleine longueur.
Amorce spécifique du gène : complémentaire à la séquence matrice, adaptée aux situations où la séquence cible est connue.
Il y a deux manières :
1. Méthode de référence interne : En théorie, l'ADNc est constitué de fragments d'ADN de différentes longueurs, le résultat de l'électrophorèse est donc un frottis. Si l'abondance d'ARN est faible, aucun produit n'apparaîtra en électrophorèse, mais cela ne signifie pas qu'aucun produit ne sera amplifié par PCR. En général, une référence interne peut être utilisée pour détecter l'ADNc. Si la référence interne a des résultats, la qualité de l'ADNc peut être essentiellement garantie (dans quelques cas, si le fragment de gène cible est trop long, il peut y avoir des exceptions).
2. S'il existe un gène connu amplifié par cette matrice, il peut être vérifié par les amorces de ce gène. L'amplification de référence interne ne signifie pas nécessairement qu'il n'y a pas de problème avec l'ADNc. Parce que la référence interne a une abondance élevée dans l'ADNc, il est facile à amplifier. Si l'ADNc est partiellement dégradé pour diverses raisons, du point de vue de la probabilité, les résultats de la PCR des gènes cibles de faible abondance seront grandement affectés. Alors que la référence interne est encore élevée en abondance, l'amplification ne sera probablement pas affectée.
Dégrader partiellement l'ARN. Détecter l'intégrité et purifier l'ARN
Les contenus en ARN de différentes espèces peuvent être différents, mais en général, l'ARN total extrait doit contenir deux bandes claires 28S et 18S en électrophorèse sur gel, et la luminosité de la première bande doit être deux fois plus élevée que celle de la seconde. La bande 5S indique que l'ARN a été dégradé et sa luminosité est proportionnelle au degré de dégradation. L'amplification réussie de la référence interne ne signifie pas qu'il n'y a pas de problème avec l'ARN, car la référence interne est en grande abondance, l'ARN peut être amplifié tant que la dégradation n'est pas sévère. L'OD260/OD280rapport d'ARN pur mesuré par spectrophotomètre doit être compris entre 1,9 et 2,1. Une petite quantité d'impureté protéique dans l'ARN réduira le rapport. Tant que la valeur n'est pas trop faible, RT ne sera pas affecté. Ce qui compte le plus pour la RT, c'est l'intégrité de l'ARN.
L'extension du gène de référence interne peut seulement indiquer que la RT a réussi, mais elle n'est pas nécessairement liée à la qualité du brin d'ADNc. Parce que les fragments de référence internes sont généralement de petite taille et d'expression élevée, ils sont plus faciles à réussir dans la transcription inverse. Cependant, la taille et l'expression du gène cible varient d'un gène à l'autre. La qualité de l'ADNc ne peut pas être jugée uniquement par référence interne, en particulier pour les fragments cibles de plus de 2 kb.
Certains échantillons ont des structures secondaires complexes, ou ont un contenu GC riche, ou sont précieux avec une faible abondance. Dans ces cas, la transcriptase inverse appropriée doit être sélectionnée en fonction de la taille du fragment cible et de l'échantillon. Pour les matrices d'ARN avec une teneur élevée en GC et une structure secondaire complexe, il est difficile d'ouvrir la structure secondaire à basse température, ou avec une transcriptase inverse commune. Pour ces matrices, la transcriptase inverse Quant peut être sélectionnée, car ses performances de transcription inverse sont évidemment meilleures que celles de la transcriptase inverse de la série M-MLV, qui peut inverser la transcription de diverses matrices d'ARN efficacement et transcrire l'ARN en premier brin d'ADNc au maximum. Lors de l'utilisation d'un kit général de transcriptase inverse, le système de 20 l ne peut efficacement transcrire que 1 g d'ARN total. Veuillez faire attention à la capacité RT maximale du kit. Si la matrice est ajoutée en excès, la transcription inverse favorisera l'ARN à forte abondance. Par conséquent, il est préférable de ne pas dépasser la capacité maximale du système.
A-1 Déterminer si l'ARN est gravement dégradé et si la RT est réussie
En général, la raison de l'échec de l'amplification de référence interne est souvent causée par une grave dégradation de l'ARN. Une autre raison possible est l'échec de la transcription inverse. La référence interne ne peut pas être utilisée comme standard pour juger de la qualité d'un simple brin d'ADNc, mais elle peut être utilisée comme standard pour juger si la transcription inverse est réussie s'il n'y a pas de problème de qualité de l'ARN. La chose la plus importante dans le processus de transcription inverse est de maintenir une température constante et un système de réaction constant afin d'améliorer l'efficacité de la réaction.
A-2 Déterminer si les amorces pour l'amplification des gènes de référence internes sont fiables et s'il y a des problèmes avec les réactifs utilisés en PCR.
Pour la quantification relative, l'ARN doit être quantifié avant la transcription inverse, ce qui est également requis dans de nombreux kits de transcription inverse, par exemple, quantifier l'entrée d'ARN à 1 g. Étant donné que l'ADNc transcrit en sens inverse est une solution mixte, comprenant de l'ARN, de l'oligo dT, de l'enzyme, du dNTP et même un petit résidu d'ADN, une déviation sera provoquée, il est donc impossible de quantifier avec précision l'ADNc. Par conséquent, la quantification de l'ARN est nécessaire. Étant donné que l'efficacité de la transcription inverse est la même parmi les différents échantillons, la quantité d'ADNc obtenue doit être la même et l'analyse quantitative peut montrer la comparaison des niveaux d'expression de différents gènes dans la même quantité d'ARN total. Lors de la réalisation d'une PCR quantitative à fluorescence relative, l'ADNc quantitatif peut ne pas être requis après la transcription inverse car le gène de référence interne peut servir de référence.
Elle est principalement liée aux gènes, et la transcription inverse d'un long fragment n'est pas réalisable pour la plupart des gènes. Premièrement, l'efficacité de la transcription inverse est bien inférieure à celle de la PCR. Deuxièmement, la région riche en GC et la structure secondaire de nombreux gènes restreignent à la fois la transcription inverse et la PCR. Enfin, la fidélité et l'efficacité d'amplification de la PCR sont difficiles à garantir en même temps. Dans le processus de transcription inverse, personne ne peut garantir l'obtention d'un long fragment pour les gènes à faible copie, en particulier en utilisant l'oligo dT. Quant au 5' UTR avec plus de GC, c'est encore plus difficile. Par conséquent, il s'agit toujours d'une méthode raisonnable pour inverser la transcription avec des amorces aléatoires, trouver les sites de clivage naturels dans le fragment cible, amplifier par segments, puis effectuer la digestion de restriction et la ligature. En général, il est difficile d'amplifier directement des fragments de plus de 2 kb, mais il n'est pas toujours impossible d'obtenir : 1. Tout d'abord, garantir l'intégrité des ARN/ARNm, et l'extraction au TRIZOL est privilégiée. Le kit 2.M-MLV RT-PCR peut être directement utilisé. Prolongez le temps de recuit et augmentez correctement le nombre de cycles dans le processus d'amplification. Alternativement, la PCR nichée peut être appliquée, ou effectuer une ou deux réactions d'abord avec une dénaturation et un temps d'extension prolongés de manière appropriée avant l'amplification PCR normale, ce qui peut aider à étendre les fragments. Faites attention à la fidélité de la polymérase. 3.Long Taq peut être utilisé en PCR pour obtenir des résultats idéaux. 4.Pour l'application d'expression de protéines, une polymérase haute fidélité doit être appliquée.
Il existe deux types de transcriptase inverse proposés par TIANGEN : Quant/King RTase et TIANScript M-MLV. La principale différence entre eux est la quantité d'entrée de modèles. Quant est une transcriptase inverse unique, qui est différente du M-MLV couramment utilisé dérivé du virus de la leucémie murine de Moloney. Quant est une nouvelle transcriptase inverse à haute efficacité exprimée de manière recombinante par ingénierie d'Escherichia coli. Quant convient à l'amplification de 50 ng-2 g d'ARN avec une activité transcriptionnelle inverse élevée et un rendement élevé. Comparé au MMLV ou à l'AMV ordinaire, la plus grande caractéristique de Quant est qu'il a une très forte affinité avec les matrices d'ARN et peut inverser les matrices complexes de transcription sans dénaturation à haute température. Pour les modèles avec un contenu GC plus élevé, l'efficacité inverse est plus élevée. Cependant, cette transcriptase inverse a une activité RNase H, ce qui peut affecter la longueur du produit d'ADNc (convient pour les matrices < 4,5 kb). Pour la transcription inverse conventionnelle, la transcriptase inverse TIANScript MMLV est recommandée. Cette RTase est une enzyme modifiée avec une activité RNase H très faible, qui convient à la synthèse d'ADNc longue (> 5 kb).
La transcription inverse en une étape et l'amplification PCR sont effectuées dans le même tube sans ouvrir le couvercle du tube entre la synthèse et l'amplification de l'ADNc, ce qui est utile pour réduire la contamination. Puisque tous les échantillons d'ADNc obtenus sont utilisés pour l'amplification, la sensibilité est plus élevée, avec un minimum de 0,01 pg d'ARN total. Pour un RTPCR réussi en une étape, des amorces spécifiques d'un gène sont généralement utilisées pour initier la synthèse d'ADNc. La méthode en deux étapes, à savoir la transcription inverse et l'amplification PCR est réalisée en deux étapes. Tout d'abord, la transcription inverse est effectuée à partir d'une matrice d'ARN pour obtenir l'ADNc, et l'ADNc obtenu est soumis à une ou plusieurs réactions PCR différentes. La méthode en deux étapes peut utiliser des oligo(dT) ou des amorces aléatoires pour guider la synthèse du premier brin d'ADNc et peut transcrire à l'envers toutes les informations d'ARNm d'un échantillon spécifique.
Catégories de produits
POURQUOI NOUS CHOISIR
Depuis sa création, notre usine a développé des produits de première classe mondiale en respectant le principe
de qualité d'abord. Nos produits ont acquis une excellente réputation dans l'industrie et une grande confiance parmi les nouveaux et les anciens clients.










