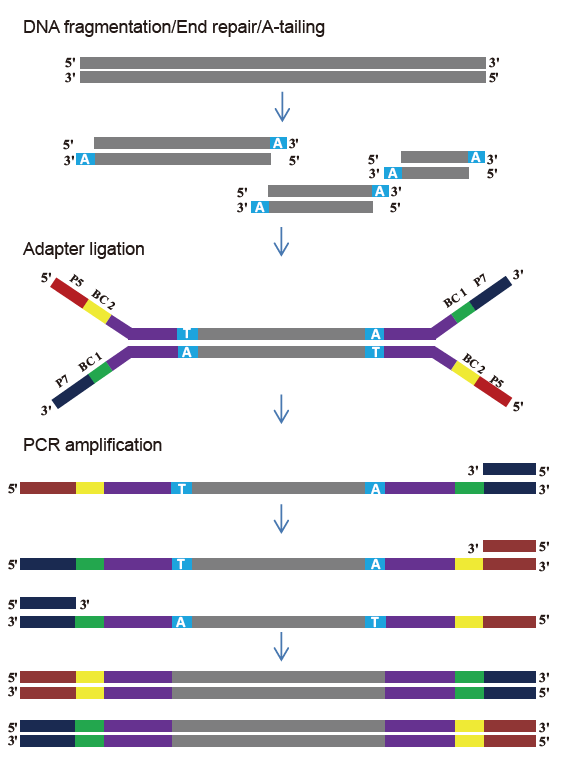
Kit de bibliothèque TIANSeq DirectFast (illumina)
Caractéristiques
■ Bonne uniformité de séquençage : Pas de biais de base du processus de fragmentation de l'ADN et du processus d'amplification PCR.
■ Efficacité de conversion élevée de la bibliothèque : la construction de la bibliothèque à haute efficacité peut être assurée pour des échantillons d'ADN de 1 ng.
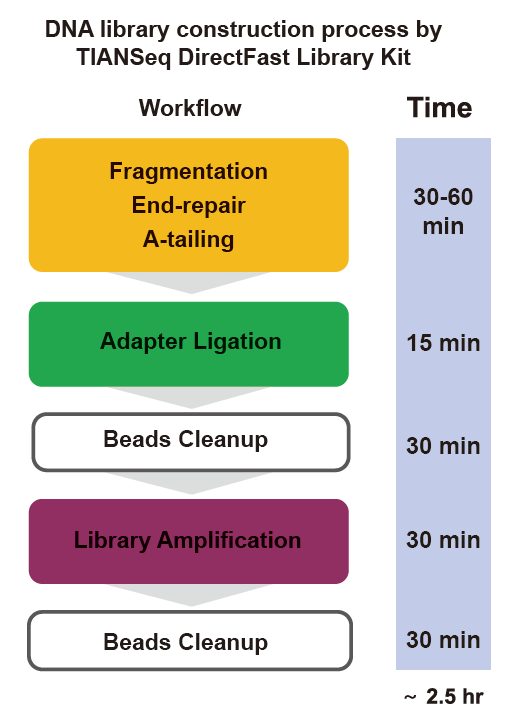
■ Opération rapide : L'ensemble du processus de construction de la bibliothèque n'a besoin que de 2,5 heures.
■ Économique : aucun instrument ni équipement spécial n'est nécessaire。
spécification
Taper: Préparation d'une banque d'ADN pour la plate-forme de séquençage à haut débit illumina
Échantillon: ADN génomique ou grand fragment d'ADN
Cible: ADN double brin
Exemple d'entrée de départ : 1 ng - 1 g
Moment de l'opération: 2,5 heures
Applications en aval : Séquençage sur la plate-forme illumina
Tous les produits peuvent être personnalisés pour ODM/OEM. Pour plus de détails,s'il vous plaît cliquez sur Service personnalisé (ODM/OEM)
Entrée d'échantillon flexible et taille fragmentée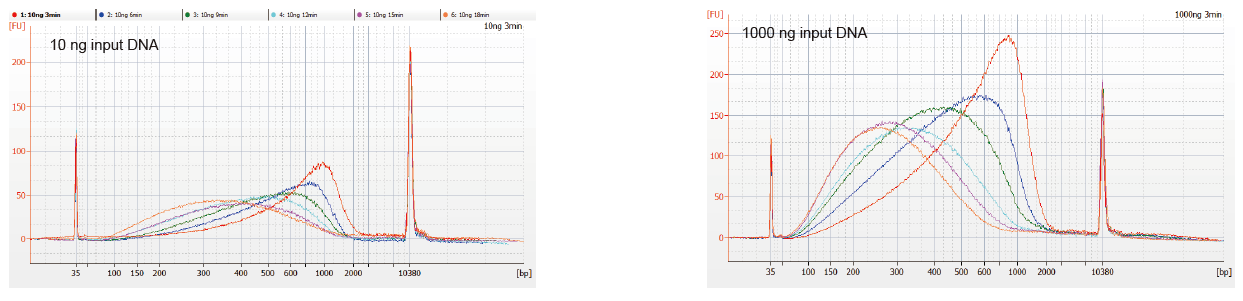 |
Figure 1. Profils de fragmentation de l'ADN de différents temps de réaction. 10 ng et 1 000 ng d'ADN ont été fragmentés à l'aide du kit TIANSeq DirectFast DNA Library. Les produits de réaction traités avec des temps de réaction différents ont été purifiés par des billes magnétiques Ampure XP 1,8 x et analysés par Angilent 2100. |
Couverture de séquençage de type Covaris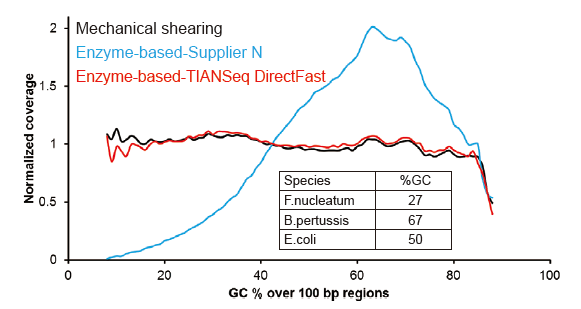 |
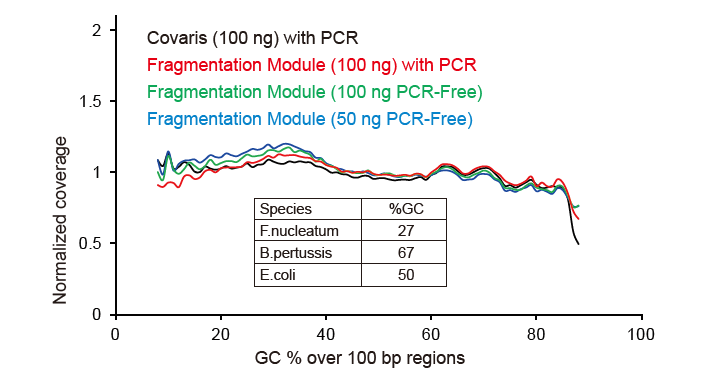
Figure 2. Comparaison de la couverture du génome de différentes méthodes de préparation de bibliothèques. Trois ADN génomiques bactériens avec des contenus GC différents sont équimolaires mixtes, et le résultat de la couverture du génome du séquençage de 100 ng de bibliothèques d'ADN mixtes utilisant ces méthodes a été comparé. Les résultats montrent que le kit TIANSeq DirectFast Library a le même effet sur la fragmentation de l'ADN que le cisaillement mécanique, et qu'il n'y a pas de biais de base pour la fragmentation. |
Pas de biais systématique pour aussi peu que 1 ng d'ADN d'entrée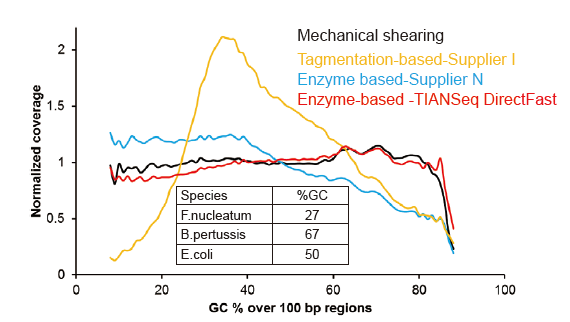 |
Figure 3. Comparaison de la couverture du génome de différentes méthodes de préparation de bibliothèques. Trois ADN génomiques bactériens avec des contenus GC différents sont équimolaires mixtes, et le résultat de la couverture du génome du séquençage de 1 ng de bibliothèques d'ADN mixtes utilisant ces méthodes a été comparé. Les résultats montrent que le kit de bibliothèque TIANSeq DirectFast a un effet de fragmentation constant avec un cisaillement mécanique, même pour une entrée d'ADN aussi faible que 1 ng, et qu'il n'y a pas de biais de base. |
| Capable de flux de travail sans PCR
|
Figure 4. Différentes entrées d'ADN génomique ont été utilisées pour construire la bibliothèque par PCR ou construction de bibliothèque sans PCR, et les résultats de couverture du génome ont été comparés. Les résultats montrent qu'avec l'opération à un tube et les étapes de construction de bibliothèque efficaces, la bibliothèque d'ADN construite avec le kit de bibliothèque TIANSeq DirectFast maintient une cohérence élevée avec le cisaillement mécanique dans la distribution de couverture de séquence de fragments pour les deux flux de travail d'enrichissement PCR sans PCR. |
Statistiques sur l'efficacité et le rendement de la construction de bibliothèques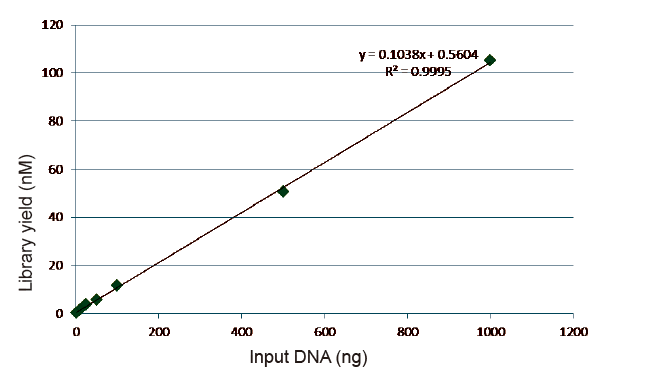 |
Figure 5. Résultats de l'analyse quantitative de l'ADN de la bibliothèque obtenu par qPCR après construction de la bibliothèque par la méthode sans PCR pour des échantillons avec différentes quantités de départ (1, 10, 25, 50, 100, 500,1000 ng). L'analyse de régression linéaire montre que le rendement de la bibliothèque a une bonne relation linéaire dans une large plage d'entrées d'échantillons. Pour une entrée d'ADN aussi faible que 1 ng, l'efficacité de la construction de la bibliothèque ne diminue pas. |
Comparaison des données de séquençage de différents produits
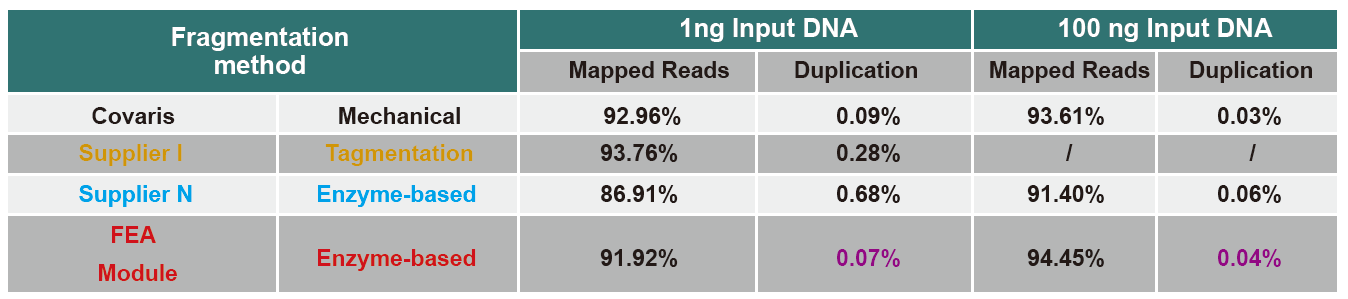
À l'heure actuelle, la technologie de séquençage à haut débit est principalement basée sur la technologie de séquençage de nouvelle génération. Comme la longueur de lecture de la technologie de séquençage de nouvelle génération est limitée, nous devons diviser la séquence complète en petites bibliothèques de fragments à séquencer. Selon les besoins des différentes expériences de séquençage, nous choisissons généralement le séquençage simple ou le séquençage double. Actuellement, les fragments d'ADN de la banque de séquençage de nouvelle génération sont généralement distribués dans la plage de 200 à 800 pb.

a) L'ADN est de mauvaise qualité et contient des inhibiteurs. Utilisez des échantillons d'ADN de haute qualité pour éviter l'inhibition de l'activité enzymatique.
b) La quantité d'échantillon d'ADN est insuffisante lors de l'utilisation d'une méthode sans PCR pour construire une bibliothèque d'ADN. Lorsque l'entrée de l'ADN fragmenté dépasse 50 ng, un flux de travail sans PCR peut être effectué de manière sélective pendant le processus de construction de la bibliothèque. Si le nombre de copies de la banque est trop faible pour être directement séquencé, la banque d'ADN peut être amplifiée par PCR après la ligature de l'adaptateur.
c) La contamination par l'ARN entraîne une quantification initiale inexacte de l'ADN Une contamination par l'ARN peut exister dans le processus de purification de l'ADN génomique, ce qui peut entraîner une quantification inexacte de l'ADN et une charge d'ADN insuffisante lors de la construction de la bibliothèque. L'ARN peut être éliminé par traitement avec la RNase.
A-1
a) Apparition de petits fragments (60 pb-120 pb) Les petits fragments sont généralement des fragments adaptateurs ou des dimères formés par des adaptateurs. La purification avec les billes magnétiques Agencourt AMPure XP permet d'éliminer efficacement ces fragments d'adaptateur et d'assurer la qualité du séquençage.
b) De gros fragments apparaissent dans la bibliothèque après amplification par PCR La taille du fragment d'ADN de la bibliothèque augmentera de 120 pb après la ligature de l'adaptateur. Si le fragment d'ADN augmente de plus de 120 pb après la ligature de l'adaptateur, cela peut être dû à une amplification anormale du fragment ou à une amplification PCR excessive. Réduire le nombre de cycles PCR peut éviter la situation.
c) Taille anormale des fragments d'ADN de la banque après ligature de l'adaptateur La longueur de l'adaptateur dans ce kit est de 60 pb. Lorsque les deux extrémités du fragment sont ligaturées aux adaptateurs, la longueur n'augmentera que de 120 pb. Lorsque vous utilisez un adaptateur autre que celui fourni par ce kit, veuillez contacter le fournisseur pour fournir des informations pertinentes telles que la longueur de l'adaptateur. Veuillez vous assurer que le flux de travail et le fonctionnement de l'expérience suivent les étapes décrites dans le manuel.
d) Taille anormale des fragments d'ADN avant la ligature de l'adaptateur La raison de ce problème peut être due à de mauvaises conditions de réaction lors de la fragmentation de l'ADN. Différents temps de réaction doivent être utilisés pour différentes entrées d'ADN. Si l'entrée d'ADN est supérieure à 10 ng, nous recommandons de choisir le temps de réaction de 12 min comme temps de départ pour l'optimisation, et la taille des fragments produits à ce moment est principalement comprise entre 300 et 500 pb. Les utilisateurs peuvent augmenter ou diminuer la longueur des fragments d'ADN pendant 2 à 4 minutes selon leurs propres besoins pour optimiser les fragments d'ADN avec la taille requise.
A-2
a) Le temps de fragmentation n'est pas optimisé Si l'ADN fragmenté est trop petit ou trop grand, veuillez vous référer aux Directives pour la sélection du temps de fragmentation fournies dans les instructions pour déterminer le temps de réaction, et utilisez ce point de temps comme contrôle, configurez en plus un système de réaction pour prolonger ou raccourcir 3 min pour faire un ajustement plus précis sur le temps de fragmentation.
A-3
Distribution de taille anormale de l'ADN après traitement de fragmentation
a) Méthode de décongélation incorrecte du réactif de fragmentation, ou le réactif n'est pas complètement mélangé après décongélation. Décongeler le réactif 5× Fragmentation Enzyme Mix sur de la glace. Une fois décongelé, mélanger le réactif uniformément en tapotant doucement le fond du tube. Ne pas vortexer le réactif !
b) L'échantillon d'ADN d'entrée contient de l'EDTA ou d'autres polluants. L'épuisement des ions sels et des agents chélatants lors de l'étape de purification de l'ADN est particulièrement important pour le succès de l'expérience. Si l'ADN est dissous dans 1 × TE, utilisez la méthode fournie dans les instructions pour effectuer la fragmentation. Si la concentration d'EDTA dans la solution est incertaine, il est recommandé de purifier l'ADN et de le dissoudre dans de l'eau déminéralisée pour une réaction ultérieure.
c) Quantification initiale inexacte de l'ADN La taille de l'ADN fragmenté est étroitement liée à la quantité d'ADN introduite. Avant le traitement de fragmentation, une quantification précise de l'ADN à l'aide de Qubit, Picogreen et d'autres méthodes est essentielle pour déterminer la quantité exacte d'ADN dans le système réactionnel.
d) La préparation du système réactionnel ne suit pas les instructions La préparation du système réactionnel fragmenté doit être effectuée sur de la glace strictement selon les instructions. Pour assurer le meilleur effet, tous les composants de la réaction doivent être placés sur de la glace et la préparation du système de réaction doit être effectuée après refroidissement complet. Une fois la préparation terminée, veuillez feuilleter ou pipeter pour bien mélanger. Ne pas vortexer !
1. Une méthode de mélange incorrecte (vortex, oscillation violente, etc.) entraînera une distribution anormale des fragments de bibliothèque (comme illustré dans la figure suivante), affectant ainsi la qualité de la bibliothèque. Par conséquent, lors de la préparation de la solution de réaction du mélange de fragmentation, veuillez pipeter doucement de haut en bas pour mélanger, ou utilisez le bout du doigt pour feuilleter et mélanger uniformément. Attention à ne pas mélanger avec le vortex.

2. L'ADN de haute pureté doit être utilisé pour la construction de la bibliothèque
■ Bonne intégrité de l'ADN : La bande d'électrophorèse est supérieure à 30 kb, sans queue
■ DE260/230 : > 1,5
■ DE260/280 : 1,7-1,9
3. La quantité d'ADN saisie doit être précise Il est suggéré d'utiliser les méthodes Qubit et PicoGreen pour quantifier l'ADN, plutôt que Nanodrop.
4. La teneur en EDTA de la solution d'ADN doit être déterminée. L'EDTA a une grande influence sur la réaction de fragmentation. Si la teneur en EDTA est élevée, une purification de l'ADN doit être effectuée avant le test suivant.
5. La solution de réaction de fragmentation doit être préparée sur de la glace. Le processus de fragmentation est sensible à la température et au temps de réaction (en particulier après l'ajout d'un activateur). Afin d'assurer l'exactitude du temps de réaction, veuillez préparer le système de réaction sur de la glace.
6. Le temps de réaction de fragmentation doit être précis Le temps de réaction de l'étape de fragmentation affectera directement la taille des produits de fragments, affectant ainsi la distribution de taille des fragments d'ADN dans la bibliothèque.
1. Quel type d'échantillon est applicable à ce kit ?
Le type d'échantillon applicable de ce kit peut être un ARN total ou un ARNm purifié avec une bonne intégrité de l'ARN. Si l'ARN total est utilisé pour construire la bibliothèque, il est recommandé d'utiliser le kit de déplétion d'ARNr (Cat#4992363/4992364/4992391) pour éliminer d'abord l'ARNr.
2. Les échantillons FFPE peuvent-ils être utilisés pour construire une bibliothèque avec ce kit ?
L'ARNm dans les échantillons FFPE sera dégradé dans une certaine mesure, avec une intégrité relativement faible. Lors de l'utilisation de ce kit pour la construction de la bibliothèque, il est recommandé d'optimiser le temps de fragmentation (raccourcir le temps de fragmentation ou ne pas effectuer de fragmentation).
3. À l'aide de l'étape de sélection de la taille fournie dans le manuel du produit, qu'est-ce qui peut provoquer une légère déviation du segment inséré ?
La sélection de la taille doit être effectuée en stricte conformité avec l'étape de sélection de la taille dans ce manuel du produit. S'il y a un écart, la raison peut être que les billes magnétiques ne sont pas équilibrées à température ambiante ou ne sont pas complètement mélangées, la pipette n'est pas précise ou le liquide est resté dans la pointe. Il est recommandé d'utiliser les pointes à faible adsorption pour l'expérience.
4. Sélection d'adaptateurs dans la construction de bibliothèques
Le kit de construction de bibliothèque ne contient pas de réactif adaptateur et il est recommandé d'utiliser ce kit avec l'adaptateur à index unique TIANSeq (Illumina) (4992641/4992642/4992378).
5. CQ de la bibliothèque
Détection quantitative de la bibliothèque : Qubit et qPCR sont utilisés pour déterminer respectivement la concentration massique et la concentration molaire de la bibliothèque. L'opération est strictement conforme au manuel du produit. La concentration de la bibliothèque répondra généralement aux exigences du séquençage NGS. Détection de la plage de distribution de la bibliothèque : Utilisation du bioanalyseur Agilent 2100 pour détecter la plage de distribution de la bibliothèque.
6. Sélection du nombre de cycle d'amplification
Selon les instructions, le nombre de cycles PCR est de 6 à 12, et le nombre de cycles PCR nécessaires doit être sélectionné en fonction de l'échantillon d'entrée. Dans les bibliothèques à haut rendement, la suramplification se produit généralement à des degrés divers, ce qui se manifeste par un pic légèrement plus grand après le pic de la plage cible dans la détection du bioanalyseur Agilent 2100, ou la concentration détectée de Qubit est inférieure à celle de qPCR. Une suramplification légère est un phénomène normal, qui n'affecte pas le séquençage de la bibliothèque et l'analyse des données ultérieure.
7. Des pointes apparaissent dans le profil de détection du bioanalyseur Agilent 2100
L'apparition de pointes dans la détection du bioanalyseur Agilent 2100 est due à la fragmentation inégale des échantillons, où il y aura plus de fragments dans une certaine taille, et cela deviendra plus évident après l'enrichissement par PCR. Dans ce cas, il est suggéré de ne pas effectuer la sélection de taille, c'est-à-dire de régler la condition de fragmentation à 94°C pendant 15 min d'incubation, où la distribution des fragments est petite et concentrée, et l'homogénéité peut être améliorée.
Catégories de produits
POURQUOI NOUS CHOISIR
Depuis sa création, notre usine a développé des produits de première classe mondiale en respectant le principe
de qualité d'abord. Nos produits ont acquis une excellente réputation dans l'industrie et une grande confiance parmi les nouveaux et les anciens clients.