Kit PCR ultra haute fidélité
Caractéristiques
■ Facile à utiliser : ce kit est fourni sous forme de prémélange 2×, et la PCR peut être effectuée en ajoutant simplement des modèles et des amorces.
■ Haute fidélité : La fidélité est 50 fois supérieure à celle de la Taq polymérase.
■ Haute spécificité : Excellentes performances de démarrage à chaud pour garantir la spécificité du produit.
■ Amplification rapide : La vitesse d'extension peut atteindre 10-15 sec/kb.
■ Forte extensibilité : jusqu'à 20 kb de fragments d'ADN peuvent être amplifiés.
■ Large applicabilité : le kit contient un amplificateur de PCR et convient à l'amplification de modèles à GC élevé et complexes.
spécification
Type : ADN polymérase haute fidélité
Vitesse d'amplification : 10-15 sec/kb
Taille des fragments : <20 ko
Applications : amplification PCR haute fidélité, clonage de gènes, amplification de matrice GC élevée, clonage de gènes de génomes complexes, amplification haute fidélité d'ADNc, détection de SNP, mutation spécifique au site, etc.
Rendement d'extraction d'ADN à partir de divers tissus végétaux :
Remarque : le rendement en ADN dépend des types d'échantillons. Tous les matériaux ci-dessus proviennent de feuilles tendres.
Tous les produits peuvent être personnalisés pour ODM/OEM. Pour plus de détails,s'il vous plaît cliquez sur Service personnalisé (ODM/OEM)
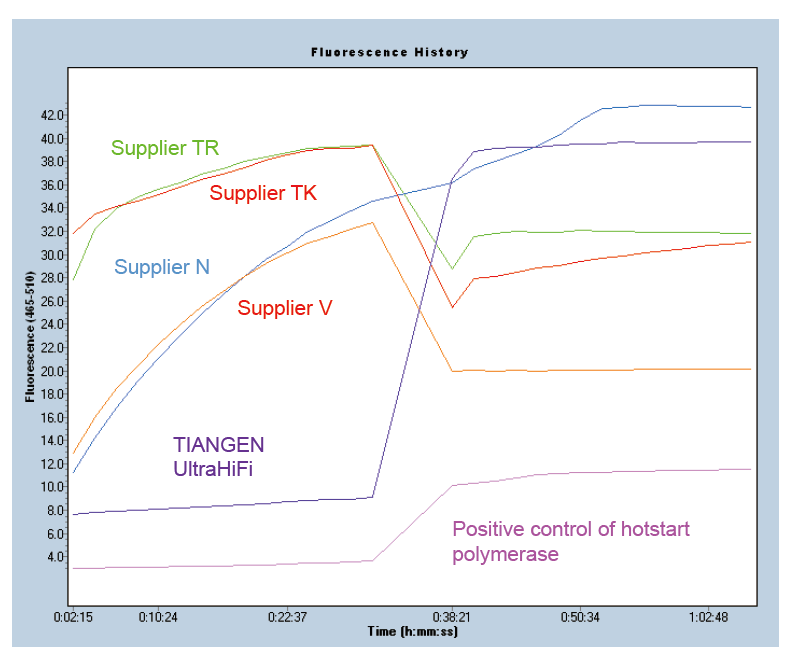 |
Démarrage à chaud pour garantir la spécificité du produit Figure 1. Ultra HiFi possède une excellente fonction de démarrage à chaud pour garantir la spécificité des produits d'amplification. La méthode des balises moléculaires a été appliquée (Ma et al., Anal Biochem, 2006). |
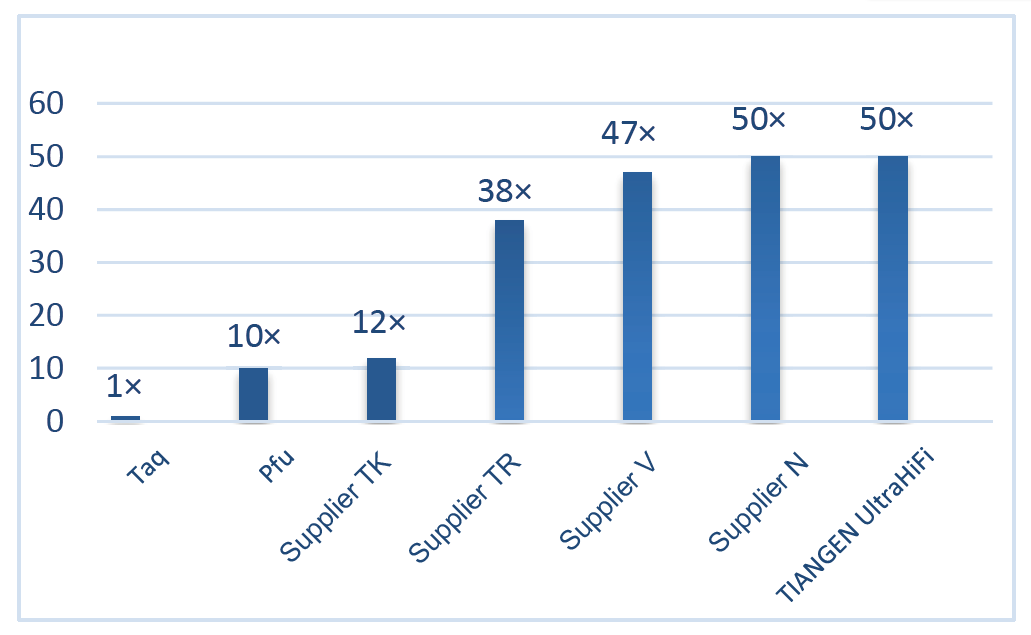 |
Excellente haute fidélité, 50 fois supérieure à la Taq Polymerase Figure 2. La fidélité de l'Ultra HiFi est 50 fois supérieure à celle de la polymérase Taq courante. La fidélité de polymérisation de la Taq polymérase (sans activité correctrice) est utilisée comme référence. |
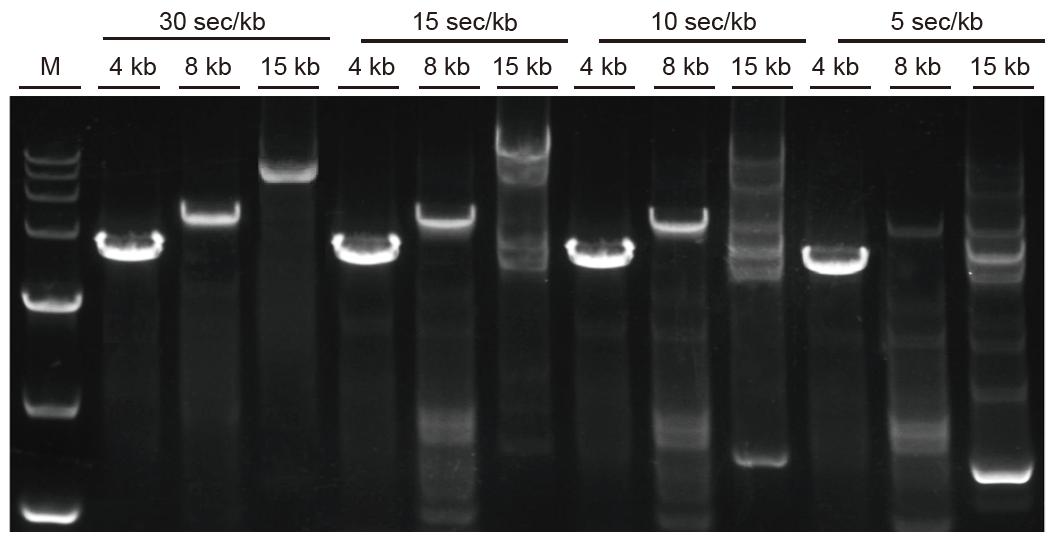 |
L'amplification rapide et les longs fragments peuvent être amplifiés rapidement Fig. 3. L'Ultra HiFi peut s'étendre jusqu'à 5 sec/kb pour des fragments inférieurs à 4 kb. Pour les fragments longs, le temps d'amplification peut être prolongé de manière appropriée. Pour les fragments de plus de 15 ko, la vitesse d'extension peut aller jusqu'à 30 s/ko. M : Marqueur TIANGEN D15000 |
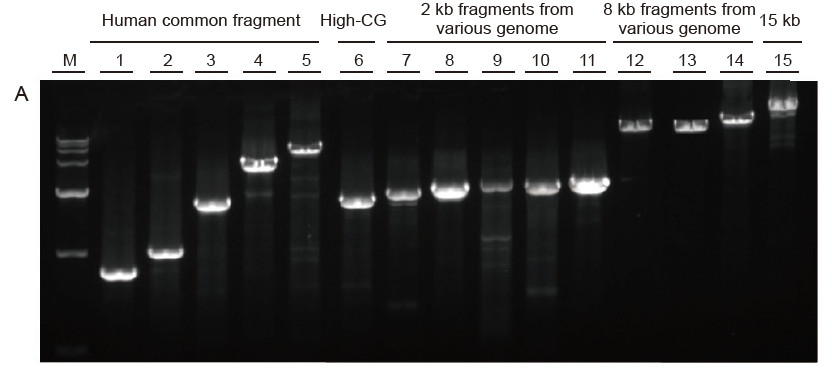 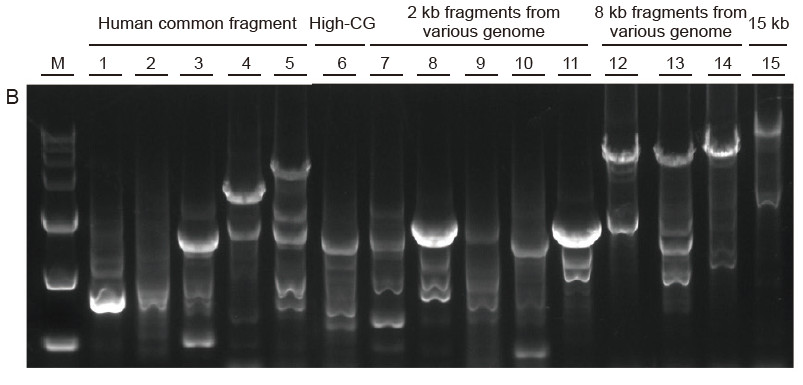 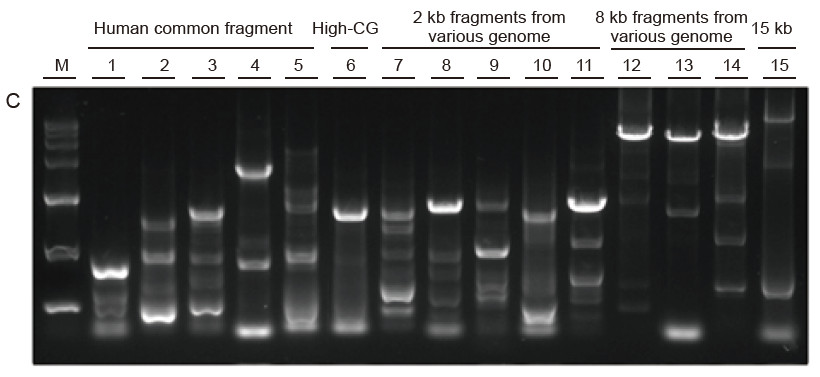 |
Forte universalité et haute spécificité, GC élevé facile à lire et longs fragments de différentes sources Figure 4. Ultra HiFi a une spécificité élevée pour assurer le taux de réussite de l'amplification et la quantité de produit pour différents types de modèles. A. Résultats de l'amplification Ultra HiFi B. Résultats de l'amplification enzymatique Hi-Fi du fournisseur K C. Résultats de l'amplification enzymatique Hi-Fi du fournisseur N M : Marqueur TIANGEN D15000 Piste 1-5. Résultats d'amplification de modèles de longueur différente : 1. 750 pb ; 2. 1 ko ; 3. 2 ko ; 4. 4 ko ; 5. 6 ko Piste 6. Résultat d'amplification d'une matrice GC élevée : 1915 pb (% GC : 70 %) ; Voie 7-11. Résultat d'amplification de modèles de 2 kb provenant de divers génomes : 7. Rat ; 8. Riz; 9. Blé; 10. Maïs ; 11. Bactéries ; Voie 12-14. Résultat d'amplification d'un fragment de 8 kb de long : 12. Riz ; 13. Maïs ; |
Modèle A-1
■ La matrice contient des impuretés protéiques ou des inhibiteurs de Taq, etc. ——Purifiez la matrice d'ADN, éliminez les impuretés protéiques ou extrayez l'ADN matrice avec des kits de purification.
■ La dénaturation de la matrice n'est pas complète ——Augmentez de manière appropriée la température de dénaturation et prolongez le temps de dénaturation.
■ Dégradation du modèle ——Repréparez le modèle.
A-2 Apprêt
■ Mauvaise qualité des amorces —— Re-synthétiser l'amorce.
■ Dégradation des amorces ——Aliquoter les amorces à haute concentration dans un petit volume pour la conservation. Éviter les congélations et décongélations multiples ou la cryoconservation à 4°C à long terme.
■ Conception incorrecte des amorces (par exemple, longueur d'amorce insuffisante, dimère formé entre les amorces, etc.) -Reconception des amorces (éviter la formation de dimère d'amorce et de structure secondaire)
A-3 mg2+concentration
■ mg2+ la concentration est trop faible ——Augmentez correctement Mg2+ concentration : Optimiser le Mg2+ concentration par une série de réactions de 1 mM à 3 mM avec un intervalle de 0,5 mM pour déterminer le Mg optimal2+ concentration pour chaque matrice et amorce.
A-4 Température de recuit
■ La température de recuit élevée affecte la liaison de l'amorce et de la matrice. ——Réduire la température de recuit et optimiser la condition avec un gradient de 2°C.
A-5 Temps de prolongation
■ Durée d'extension courte——Augmente la durée d'extension.
Phénomènes : Les échantillons négatifs montrent également les bandes de séquence cible.
A-1 Contamination de la PCR
■ Contamination croisée de la séquence cible ou des produits d'amplification —— Veillez à ne pas pipeter l'échantillon contenant la séquence cible dans l'échantillon négatif ou à ne pas les renverser hors du tube de centrifugation. Les réactifs ou l'équipement doivent être autoclavés pour éliminer les acides nucléiques existants, et l'existence d'une contamination doit être déterminée par des expériences de contrôle négatif.
■ Contamination des réactifs ——Aliquoter les réactifs et conserver à basse température.
A-2 Premierr
■ mg2+ la concentration est trop faible ——Augmentez correctement Mg2+ concentration : Optimiser le Mg2+ concentration par une série de réactions de 1 mM à 3 mM avec un intervalle de 0,5 mM pour déterminer le Mg optimal2+ concentration pour chaque matrice et amorce.
■ Conception d'amorce incorrecte et la séquence cible présente une homologie avec la séquence non cible. —— Re-conception des amorces.
Phénomènes : Les bandes d'amplification PCR ne correspondent pas à la taille attendue, qu'elles soient grandes ou petites, ou parfois des bandes d'amplification spécifiques et des bandes d'amplification non spécifiques se produisent.
A-1 Apprêt
■ Mauvaise spécificité d'amorce
——Amorce de refonte.
■ La concentration d'amorce est trop élevée ——Augmentez correctement la température de dénaturation et prolongez le temps de dénaturation.
A-2 mg2+ concentration
■ Le magnésium2+ la concentration est trop élevée ——Réduire correctement la concentration de Mg2+ : Optimiser la concentration de Mg2+ concentration par une série de réactions de 1 mM à 3 mM avec un intervalle de 0,5 mM pour déterminer le Mg optimal2+ concentration pour chaque matrice et amorce.
A-3 Polymérase thermostable
■ Quantité d'enzyme excessive ——Réduire la quantité d'enzyme de manière appropriée à des intervalles de 0,5 U.
A-4 Température de recuit
■ La température de recuit est trop basse ——Augmentez la température de recuit de manière appropriée ou adoptez la méthode de recuit en deux étapes
Cycles de PCR A-5
■ Trop de cycles PCR ——Réduisez le nombre de cycles PCR.
A-1 Apprêt——Mauvaise spécificité ——Re-concevoir l'amorce, changer la position et la longueur de l'amorce pour améliorer sa spécificité; ou effectuer une PCR nichée.
A-2 ADN modèle
——La matrice n'est pas pure ——Purifiez la matrice ou extrayez l'ADN avec des kits de purification.
A-3 mg2+ concentration
——Mg2+ la concentration est trop élevée ——Réduire correctement le Mg2+ concentration : Optimiser le Mg2+ concentration par une série de réactions de 1 mM à 3 mM avec un intervalle de 0,5 mM pour déterminer le Mg optimal2+ concentration pour chaque matrice et amorce.
A-4 dNTP
——La concentration de dNTP est trop élevée ——Réduire la concentration de dNTP de manière appropriée
A-5 Température de recuit
——Température de recuit trop basse ——Augmenter la température de recuit de manière appropriée
A-6 Cycles
——Trop de cycles ——Optimiser le nombre de cycles

La première étape consiste à choisir la polymérase appropriée. La polymérase Taq régulière ne peut pas relire en raison du manque d'activité de l'exonucléase 3'-5', et le mésappariement réduira considérablement l'efficacité d'extension des fragments. Par conséquent, la polymérase Taq normale ne peut pas amplifier efficacement les fragments cibles de plus de 5 kb. La polymérase Taq avec une modification spéciale ou une autre polymérase haute fidélité doit être sélectionnée pour améliorer l'efficacité d'extension et répondre aux besoins d'amplification de fragments longs. De plus, l'amplification de longs fragments nécessite également un ajustement correspondant de la conception de l'amorce, du temps de dénaturation, du temps d'extension, du pH du tampon, etc. Habituellement, les amorces avec 18-24 pb peuvent conduire à un meilleur rendement. Afin d'éviter d'endommager la matrice, le temps de dénaturation à 94°C doit être réduit à 30 secondes ou moins par cycle, et le temps d'élévation de la température à 94°C avant l'amplification doit être inférieur à 1 min. De plus, le réglage de la température d'extension à environ 68°C et la conception du temps d'extension en fonction de la vitesse de 1 kb/min peuvent garantir une amplification efficace de longs fragments.
Le taux d'erreur de l'amplification PCR peut être réduit en utilisant diverses ADN polymérases avec une haute fidélité. Parmi toutes les ADN polymérases Taq trouvées jusqu'à présent, l'enzyme Pfu a le taux d'erreur le plus bas et la fidélité la plus élevée (voir tableau ci-joint). En plus de la sélection d'enzymes, les chercheurs peuvent réduire davantage le taux de mutation PCR en optimisant les conditions de réaction, notamment en optimisant la composition du tampon, la concentration de polymérase thermostable et en optimisant le nombre de cycles de PCR.
Catégories de produits
POURQUOI NOUS CHOISIR
Depuis sa création, notre usine a développé des produits de première classe mondiale en respectant le principe
de qualité d'abord. Nos produits ont acquis une excellente réputation dans l'industrie et une grande confiance parmi les nouveaux et les anciens clients.